Príbeh o úspechoch zákazníkov:
Rýchle sekvenovanie kompletného genómu (rWGS)
Špecifikácie projektu
Projekt rWGS sekvenovania realizovaný ako “proof-of-concept experiment” bol navrhnutý v spolupráci s tímom Prof. Milana Maceka pôsobiaceho na 2. Lekárskej Fakulte Univerzity Karlovy a FN Motol. Trio vzoriek - rodičia s probandom, ktorému bol diagnostikovaný Mayer-Rokitansky-Küster-Hauserov syndróm (MRKH) - boli vybrané tímom Prof. Maceka, s cieľom ukázať efektivitu WGS v porovnaní s panelovou a/alebo WES analýzou.
Celý proces bol navyše vysielaný počas konferencie DNA diagnostika 2023, ktorá sa konala 20. a 21. marca 2023 v Lipne.
Úvod
Rýchle sekvenovanie kompletného genómu (rWGS) a jeho následná analýza, sa nedávno stalo prelomovým prístupom najmä v prípadoch, kedy je rýchlosť komplexnej genomickej analýzy kľúčová. Ilustračným príkladom môže byť testovanie pediatrických/novorodeneckých pacientov, a to hlavne z dôvodu až 40% úmrtnosti novorodencov spojenej so zriedkavým genetickým ochorením s extrémne rýchlym priebehom 1-4. V klinickej praxi môže rWGS, v porovnaní s inými diagnostickými prístupmi (Tabuľka 1), vďaka rýchlej identifikácie kauzálnych genetických variantov (vrátane komplexných štrukturálnych variantov) umožniť zlepšenie liečby alebo určenie prognózy priebehu ochorení 5.
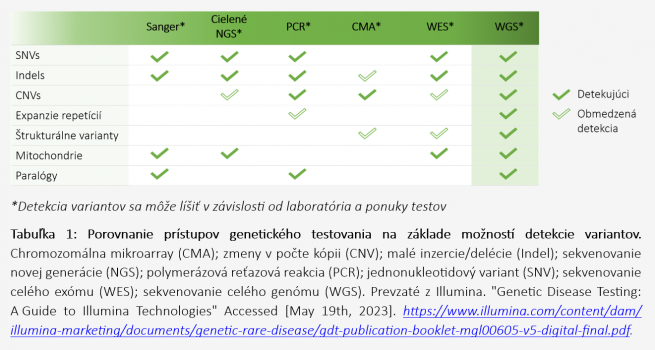
rWGS sa taktiež stáva zlatým štandardom v personalizovanej diagnostike, pretože v porovnaní s jeho prínosmi, má veľmi nízku cenu, pričom včasnou a presnou idnetifikáciou kauzálnych variant pomáha mimo iného aj znížiť náklady na celkovú starostlivosť 6.
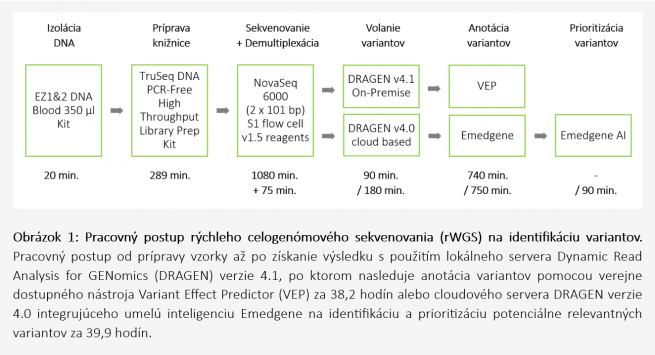
V spolupráci s Inštitútom aplikovaných biotechnológií a.s. (IAB) predstavujeme protokol rýchleho WGS poskytujúceho vysokokvalitné dáta, v ktorom sa spájajú technológie a nástroje NGS spoločností QIAGEN a Illumina. Optimalizovaný NGS protokol kombinuje najmodernejšie produkty a prístupy izolácie DNA, prípravy knižníc, sekvenovania a analýzy sekvenačných údajov s následnou interpretáciou dát, vrátane anotácie variantov. Vďaka konzultácii výsledov s profesorom Macekom, bolo možné uskutočniť kompletnú diagnostiku probanda a jeho rodičov už za 39,9 hodín (Obrázok 1).
Metódy
Purifikácia DNA: Už počas úvodného kroku pracovného postupu sa čas spracovania vzorky dramaticky skrátil využitím automatického purifikátora EZ2 Connect (QIAGEN) fungujúceho na báze magnetických guličiek. Vysokokvalitná genómová DNA (gDNA) bola izolovaná pomocou súpravy EZ1&2 DNA Blood 350 µl Kit (QIAGEN) v priebehu 20 minút z malého objemu 3 vzoriek krvi probanda a jeho rodičov (matka, otec, proband).
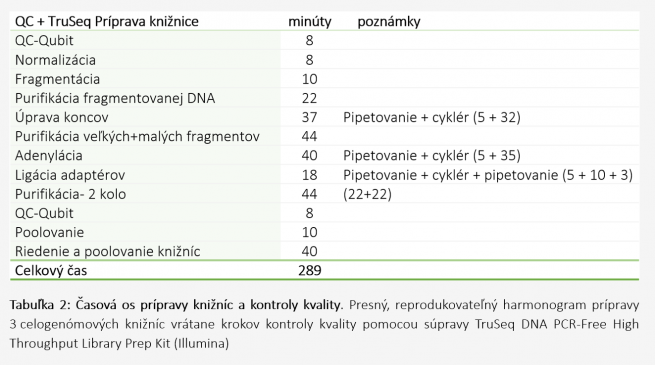
Príprava knižnice: Celogenómové knižnice so vstupnou koncentráciou gDNA 1050 ng boli pripravené a nariedené /vrátane všetkých krokov kontroly kvality/ (Tabuľka 2) za 289 minút pomocou súpravy TruSeq DNA PCR-Free High Throughput Library Prep Kit (Illumina). Protokol PCR-Free predstavuje vynikajúce riešenie pre ťažko sekvenovateľné regióny DNA, ako sú oblasti bohaté na GC páry, promótory alebo repetície. Použitie mechanickej fragmentácie navyše znižuje riziko vytvárania artefaktov a medzier v knižniciach.
Sekvenovanie: rWGS sekvenovanie bolo vykonané na prístroji NovaSeq 6000 s S1 flowcell (2x101 bp) (Illumina). Sekvenačný beh sa úspešne ukončil za 18 hodín s výsledným objemom dát: 464,92 Gb, pri 92,57 % čítaní pokrytých viac ako 35× a 94,66 % čítaním so skóre kvality Phred nad 30 (Q30). Všetky tri celogenómové knižnice spĺňali požadované kritérium - 800 miliónov čítaní z oboch koncov (Tabuľka 3).
.png)
Analýza dát: Demultiplexovanie sekvenčných dát a konverzia CBCL na FASTQ bolo ukutočnené prostredníctvom cloudového servera BaseSpace Sequence Hub (Illumina) za 75 minút. Na následnú sekundárnu analýzu boli použité dva rôzne prístupy.
Prvý prístup využil lokálny server Dynamic Read Analysis for GENomics (DRAGEN) On-Premise Server v4.1 (Illumina) s hg38 Alignment a Variant Calling. Na anotáciu identifikovaných variantov bol použitý voľne dostupný anotačný nástroj Variant Effect Predictor (VEP) (Ensembl). Vďaka absencii limitov internetového pripojenia a plnej kontrole nad prioritami spracovania sekvenačných dát trvala analýza ~ 14 hodín. V druhom prístupe bol využitý cloudový systém DRAGEN v4.0 a Emedgene (Illumina), ktoré dokončili anotáciu variantov za ~15,5 hodín. Dlhšie trvanie analýzy je však zanedbateľné, pretože klinickí špecialisti pracujú len s niekoľkými klinicky relevantnými variantmi predvybranými umelou inteligenciou Emedgene a nie so stovkou netriedených anotovaných variantov.
V tomto prípade umelá inteligencia Emedgene do užšieho výberu zaradila 7 najpravdepodobnejších patologických variantov a 63 ďalších kandidátnych variantov, ktoré boli zaslané na hodnotenie laboratórnym diagnostikom z 2. Lekárskej Fakulty Univerzity Karlovy a FN Motol. Na hlbšie preskúmanie jednotlivých prípadov poskytuje Emedgene rýchle nastavenia filtrovania variantov, ktoré môžu laboratórni diagnostici alebo iní užívatelia aplikovať na zobrazenie exónových a intrónových variantov v kandidátnych génoch alebo sa zamerať na varianty s očakávanou segregáciou, vo svojich súkromných účtoch v Emedgene.
Zhrnutie
Táto stručná správa popisuje prielomový pracovný postup rWGS, ktorý integruje všetky metodiky digitálnej genomiky vrátane umelej inteligencie do komplexného personalizovaného diagnostického prístupu. Hlavným cieľom bolo poskytnúť prioritné anotované varianty v efektívnom časovom rámci za menej ako 40 hodín od odberu vzorky. Náš inovatívny pracovný postup rWGS založený na technológiach spoločností QIAGEN a Illumina, ktorý kombinuje presnosť, rýchlosť a výnimočnú analytickú komplexnosť, posúva personalizovanú diagnostiku na úplne novú úroveň a otvára cestu širšiemu využitiu ľudskej genetiky v klinickej praxi. Prezentované výsledky dokazujú, že rWGS predstavuje najmodernejšie riešenie pre presnú diagnostiku, najmä vďaka rýchlej detekcii kauzálnych variantov genetických porúch, a to aj v náročných prípadoch, v ktorých iné diagnostické nástroje zaostávajú.
Veľké poďakovanie patrí tímom prof. Milana Maceka, IAB a našim kolegom z GeneTiCA Group, ktorí sa spoločne na celom projekte podielali!
_______________________________________________________________________________________
1. Weiner J, Sharma J, Lantos J, Kilbride H. How infants die in the neonatal intensive care unit: trends from 1999 through 2008. Arch Pediatr Adolesc Med. 2011;165(7):630-634. doi:10.1001/archpediatrics.2011.102
2. Gunne E, McGarvey C, Hamilton K, Treacy E, Lambert DM, Lynch SA. A retrospective review of the contribution of rare diseases to paediatric mortality in Ireland. Orphanet J Rare Dis. 2020;15(1):311. doi:10.1186/s13023-020-01574-7Weiner
3. Berry MA, Shah PS, Brouillette RT, Hellmann J. Predictors of mortality and length of stay for neonates admitted to children's hospital neonatal intensive care units. J Perinatol. 2008;28(4):297-302.
4. Arth AC, Tinker SC, Simeone RM, Ailes EC, Cragan JD, Grosse SD. Inpatient Hospitalization Costs Associated with Birth Defects Among Persons of All Ages - United States, 2013. MMWR Morb Mortal Wkly Rep. 2017;66(2):41-46.
5. Petrikin JE, Cakici JA, Clark MM, et al. The NSIGHT1-randomized controlled trial: rapid whole-genome sequencing for accelerated etiologic diagnosis in critically ill infants. NPJ Genom Med. 2018;3:6. doi:10.1038/s41525-018-0045-8
6. Farnaes L, Hildreth A, Sweeney NM, et al. Rapid whole-genome sequencing decreases infant morbidity and cost of hospitalization. NPJ Genom Med. 2018;3:10. doi:10.1038/s41525-018-0049-4







3.png)




.png)

1.png)

1.png)

